Eiweiße in drei Dimensionen sehen und drehen
-
 <ic:message key='Bild vergrößern' />
<ic:message key='Bild vergrößern' />
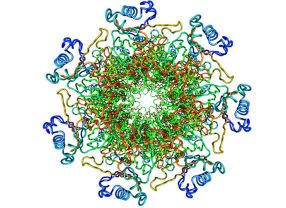
- Der Enzymkomplex des Bakteriums "Thermoplasma Acidophilum" ist für das Recycling zuständig. Wie hier im Programm Ballview zu sehen, besteht er aus 55.0000 Atomen. Quelle: Ballview
03.03.2010 -
Eiweiße sind die Arbeitstiere der Zelle. Es gibt viele hundert verschiedene Modelle, die alle eines gemeinsam haben: Sie bestehen aus Aminosäurenketten, die sich allerdings bei jedem Eiweiß in unterschiedlicher Manier falten und miteinander verbinden. Wie das komplexe Gebilde aussieht, das am Ende entsteht, ist wichtig für die Forschung: Denn die Form bestimmt bei Eiweißen die Funktion. Bioinformatiker in Saarbrücken und Tübingen haben jetzt eine Software weiterentwickelt, die Eiweiße, aber auch andere Zellmoleküle oder Viren dreidimensional darstellen kann. Mit dem frei verfügbaren Programm "BALLview" können Kollegen leichter neue Angriffspunkte für Therapien finden, hoffen die Forscher. Sie stellen die erweiterte Version zum ersten Mal auf der Computermesse Cebit vor, die noch bis zum 6. März in Hannover stattfindet.
Das größte menschliche Eiweiß Titin besteht aus über 30.000 Aminosäuren. Seine Struktur genau zu kennen ist bedeutsam für die Medizin. Titin ist im Muskel dafür zuständig, dass der sich nach einer Dehnung wieder zurückstellt. Bei einem derartig großen Molekül stößt die zweidimensionale Darstellung allerdings schnell an ihre Grenzen. Eine dreidimensionale Abbildung ist gefragt. Forscher am Max-Planck-Institut für Informatik in Saarbrücken entwickelten vor Jahren deshalb das Programm "Biochemical Algorithms Library" oder kurz BALLview.
| BALLview |
Mit dem Programm "Biochemical Algorithms Library" oder kurz Ballview können Biomoleküle dreidimensional betrachtet werden. Das Programm gibt es kostenlos auf der Webseite. |
Mit der Software können komplizierte Moleküle und deren physikalische Eigenschaften, aber auch umfangreiche biologische Systeme wie etwa Viren berechnet und visualisiert werden. Das Programm ist nicht nur kostenlos verfügbar, sondern darüber hinaus auch als Open-Source-Software angelegt. Jeder Programmierer kann also den zugrundeliegenden Programmcode einsehen und weiterentwickeln. Drei Forscherteams an den Zentren für Bioinformatik in Saarbrücken und Tübingen sind nun einige entscheidende Verbessrungen gelungen. Ballview ist nun wirklich 3D-fähig.
Dieser mit BALLview erzeugte Film zeigt die Untereinheit eines bakteriellen Ribosoms in 3D.Quelle: BALLview
Dreidimensionale Eiweiße mit der Spacemouse bewegen
Damit Forscher sich noch besser die räumlichen Strukturen der Moleküle vorstellen können, werden diese jetzt auch stereoskopisch dargestellt. Dabei werden zwei Bilder an einer Leinwand so übereinander gelegt, dass der Betrachter sie durch eine 3-D-Brille mit Tiefenwahrnehmung sehen kann. Er erhält dadurch einen realistischen räumlichen Eindruck und kann die Eiweiße oder Viren zudem direkt an der Leinwand verschieben, in einzelne Bereiche hineinzoomen und diese dann bearbeiten.
| Mehr zum Thema auf biotechnologie.de |
News: Schutzengel für Eiweiße in 3D nachgebaut Förderbeispiel: Zellverbände in 3D unter die Lupe nehmen News: Biobrowser- Kleine Eiweiße ganz groß Biotechnologie.tv: Ungewöhnliche Einblicke in 3D |
Dabei kommen moderne Eingabegeräte wie die 3-D-Spacemouse zum Einsatz, mit der man in virtuellen Umgebungen Objekte bewegen kann. Auch das so genannte Headtracking, das die Kopfbewegungen des Anwenders über Infrarotsensoren erfasst, hilft bei der Steuerung. Die Saarbrücker Bioinformatiker stellen die neueste BaLLview-Version erstmals auf der Computermesse Cebit, vor, die vom 2. bis 6. März in Hannover läuft, und zwar am saarländischen Forschungsstand in Halle 9.
| Die BALLview-Entwickler |
Arbeitsgruppe von Andreas Hildebrandt am Zentrum für Bioinformatik der Universität des Saarlandes Arbeitsgruppe von Philipp Slusallek am Deutschen Forschungszentrum für Künstliche Intelligenz Arbeitsgruppe von Oliver Kohlbacher an der Universität Tübingen |
Licht und Schatten in der Eiweißwelt
Eine weitere Neuerung ist die Möglichkeit, die räumlichen Strukturen der Moleküle mit Licht, Schatten und Spiegelungen darzustellen. Das dazu notwendige sogenannte Ray-Tracing kam bisher nur an zweidimensionalen Bildschirmen zum Einsatz. Entwickelt hat es Andreas Hildebrandt, der eine Forschergruppe am Zentrum für Bioinformatik und dem Intel Visual Computing Institute der Universität des Saarlandes leitet. Auch die Medikamentenentwicklung dürfte von der kontinuierlichen Verbesserung der Ballview-Software profitieren. Da im Medikamentendesign viele Forschergruppen auf der ganzen Welt zusammenarbeiten, haben die Saarbrücker Informatiker die Möglichkeit geschaffen, dreidimensionale Darstellungen über das Internet auszutauschen und anzuzeigen. Die dafür notwendige 3-D-Technologie für das Internet, genannt "XML3D", wurde von einem Forscherteam um Philipp Slusallek am Intel Visual Computing Institute der Universität des Saarlandes und dem Deutschen Forschungszentrum für Künstliche Intelligenz entwickelt. Durch eine Erweiterung von gewöhnlichen Webbrowser können damit komplexe dreidimensionale Graphiken verarbeitet werden. Die Integration dieser Technologie in das Ballview-Programm ermöglicht Forschergruppen das gemeinsame Arbeiten an 3D-Biomolekülen auch über weite Distanzen hinweg.