Wochenrückblick KW 32
Rückblick auf Kalenderwoche 32
Für den Zeitraum vom 5. August bis 12. August 2013 hat biotechnologie.de für Sie die wichtigsten Nachrichten aus der Biotech-Branche zusammengestellt.
3D-Ansicht der Netzhaut
-
 <ic:message key='Bild vergrößern' />
<ic:message key='Bild vergrößern' />
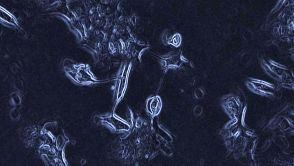
- 3D-Rekonstruktion von etwa 1000 Nervenzellen und ihren Verschaltungen in einem Netzhaut-Stück einer Maus. Quelle: MPI für medizinische Forschung / Isensee & Kuhl
Max-Planck-Forscher haben mit der Unterstützung von 200 Studenten die Nervenverbindungen einer Netzhaut kartiert und in 3D abgebildet.
Rund vier Jahre verbrachten die Forscher mit der Datenanalyse, um detailreiche 3D-Aufnahmen anzufertigen und knapp eine halbe Million Verbindungen zwischen Nervenzellen der Retina zu kartieren. Das menschliche Gehirn besitzt rund 100 Milliarden Nervenzellen und jede ist über tausende Kontakte mit anderen Zellen verbunden. Um den Aufbau des Hirnes und die Verzweigungen der Nervenbahnen genauer zu studieren, untersuchen manche Forscher das Gehirn in hauchdünnen Scheiben (mehr...). Andere wiederum fotografieren schockgefrorene Zellen aus allen Richtungen um daraus 3D-Bilder zu generieren (mehr...). Wissenschaftler der Max-Planck-Institute für medizinische Forschung in Heidelberg und Neurobiologie in Martinsried, sowie US-Kollegen des MIT setzten auf eine andere Methode: Sie prüften elektronenmikroskopischen Bilder eines Stückes einer Netzhaut und verfolgten in mühsamster Kleinarbeit einzelne Nervenverbindungen über lange Strecken.
| Mehr zum Thema auf biotechnologie.de |
News: Mit Gentherapie gegen Netzhautdegeneration Wochenrückblick: Virtueller Gehirn-Atlas aus hauchdünnen Schnitten |
"Wir brauchten ungefähr einen Monat um die Daten zu gewinnen und vier Jahre um sie zu analysieren", so der Erstautor der Studie Moritz Helmstaedter. Obwohl der untersuchte Netzhautwürfel gerade einmal einen Zehntel Millimeter Kantenlänge hatte, kamen darin knapp 1000 Nervenzellen mit rund einer halben Million Verbindungen vor. Um diese zu analysieren, setzten die Forscher nicht nur auf neueste Technik, sondern vor allem auf die Mithilfe von 200 Studenten. Diese fällten letztlich die Entscheidungen über reale und falsche Abzweigungen in den neuronalen Verdrahtungen. Die im Fachjournal Nature (2013, Online-Veröffentlichung) publizierte Arbeit verschlang somit allein rund 20.000 Arbeitsstunden. Die Fleißarbeit hat sich gelohnt: Bereits dieser vergleichsweise kleine Einblick ins Gehirn brachte sowohl einen neuen Zelltyp ans Licht, als auch Verschaltungen, die bestimmte Reaktionen einzelner Netzhautzellen erklären könnten. "Diese Ergebnisse zeigen uns, dass wir auf dem richtigen Weg sind, obwohl wir mit dieser Arbeit gerade einmal 0,1 Prozent der Netzhaut einer Maus analysiert haben", so Helmstaedter. Für eine ähnliche Entschlüsselung der Verdrahtung eines ganzen Mäusegehirns, wären allerdings mehrere Milliarden Arbeitsstunden nötig. Trotzdem hat Winfried Denk vom MPI dieses Ziel fest vor Augen: "Ich bin davon überzeugt, dass wir den automatisierten Prozess, den wir auch für das Netzhautstück verwendet haben, so skalieren können, dass man damit ein ganzes Mäusegehirn dreidimensional abbilden kann. Auch wenn wir dazu ein oder zwei Jahre durchgehend Daten aufnehmen müssen." Möglich soll dies auch durch viele freiwillige Helfer werden, so Moritz Helmstaedter: „Noch in diesem Jahr wollen wir mit dem Spiel Brainflight online gehen, in dem Internetnutzer auf der ganzen Welt Nervenbahnen nachfliegen und Punkte sammeln können.“
© biotechnologie.de/al
Wilex-Wirkstoff in klinischer Entwicklung
-
 <ic:message key='Bild vergrößern' />
<ic:message key='Bild vergrößern' />
- Die Wilex AG erhielt im Rahmen der m4-Spitzencluster-Initiative eine BMBF-Förderung in Höhe von 2,6 Millionen Euro für die Entwicklung von WX-037. Quelle: Jetti Kuhlemann / pixelio.de
Die Münchener Wilex AG bringt in Großbritannien den Wirkstoff WX-037 in die klinische Entwicklung.
An der unverblindeten Studie nehmen zunächst Patienten teil, für deren fortgeschrittene solide Tumore es keine wirksame Standardtherapie mehr gibt. Bei unverblindeten Studien ist Teilnehmern und Prüfärzten bekannt, welcher Proband das Testmedikament und welcher gegebenenfalls das Kontroll-Placebo erhält. Ziel der Studie ist es, Sicherheit, Pharmakokinetik, Pharmakodynamik und klinische Aktivität eines sogenannten PI3K-Inhibitors zu untersuchen. In einem zweiten Schritt soll das Präparat auch an Patienten mit einem deregulierten PI3K-Signalweg getestet werden. WX-037 blockiert den Phospatidylinositol-3-Kinase-Signalweg, eine Signalkaskade, die in vielen Tumorzellen mutiert ist.
| Mehr zum Thema auf biotechnologie.de |
Wochenrückblick: Wilex und Agennix: Entlassungen nach Fehlschlägen Wochenrückblick: Wilex: Nierenkrebsmittel Rencarex scheitert in Zulassungsstudie |
Die Probanden erhalten WX-037 in Kombination mit einem weiteren Wirkstoffkandidaten von Wilex. Die Rechte an der Substanz übernahm Wilex im Rahmen einer strategischen Partnerschaft 2009 vom belgischen Pharmakonzern UCB. Mit dem Projekt beteiligte sich das Münchner Biotech-Unternehmen an der Initiative „m4 Personalisierte Medizin und zielgerichtete Therapien” des Münchner Spitzenclusters m4. Vom Bundesforschungsministerium erhält Wilex eine Förderung von insgesamt bis zu 2,6 Millionen Euro für die präklinische und klinische Entwicklung von WX-037. Auch Lizenzgeber UCB scheint die weitere Entwicklung seiner Wirkstoffe genau weiter zu verfolgen. Erst vor wenigen Wochen hatte UCB die Rechte an einem Antikörperprogramm aus dem präklinischen Portfolio von Wilex zurückerworben, welches der Konzern ursprünglich 2009 an das Münchner Unternehmen abgegeben hatte. UCB kann das Immunmolekül nun in sämtlichen Indikationen außerhalb der Onkologie weiterentwickeln, während Wilex die Rechte für die Weiterentwicklung in onkologischen Indikationen beibehält. Wilex erhielt im Gegenzug eine Erstattung für die bisher entstandenen Entwicklungskosten sowie die Zusage für weitere meilensteinabhängige Prämien und Umsatzbeteiligungen.
© biotechnologie.de/bk
Kompletter Proteinkatalog für Tumorzelllinie
-
 <ic:message key='Bild vergrößern' />
<ic:message key='Bild vergrößern' />
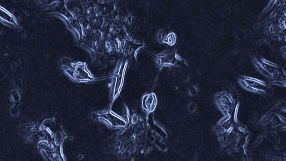
- Auch andere Versuchszelllinien nahmen die Münchener Wissenschaftler genauer unter die Lupe. Hier die Lungenkrebszellen der Linie HCC827. Quelle: K. Kramer/TUM
Münchener Krebsforscher haben sämtliche Proteine der Tumorzelllinie NCI-60 katalogisiert und neue Erkenntnisse über die Wirksamkeit von Medikamenten gewonnen.
Auf der Suche nach Ursachen, die aus einer gewöhnlichen Körperzelle eine Krebszelle werden lassen, haben sich Krebsforscher bisher auf die Ebene der Gene konzentriert. Weil es jedoch letztendlich die Proteine sind, die die Umwandlung vollziehen, haben die Münchener Wissenschaftler nun das Proteom der Tumorzellen analysiert. Dabei identifizierten die Onkologen über 10.000 verschiedene Proteine in Krebszellen und fanden Hinweise darauf, warum Arzneien bei verschiedenen Patienten unterschiedliche Wirkungen zeigen. Über die Ergebnisse ihrer Studie berichten die Forscher im Fachjournal Cell (2013, Online-Veröffentlichung).
| Mehr zum Thema auf biotechnologie.de |
Wochenrückblick: Neue Proteom-Datenbank vorgestellt |
Die Tumor-Zelllinie NCI-60 (von National Cancer Institute, USA) wird weltweit für Versuche genutzt, da sie die häufigsten Tumorerkrankungen repräsentiert. „Bisher war das Proteom der NCI-60-Zelllinen weitgehend unbekannt – dabei sind es die Proteine, die den Unterschied zwischen einer gesunden und einer Tumorzelle ausmachen“, sagt Amin Gholami, Bioinformatiker an der Technischen Universität München. Weil sich weitgehend alle Tumormedikamente gegen zelluläre Proteine richten, sei es einfacher neue Angriffspunkte für Medikamente zu finden, wenn das Proteom bekannt sei, ergänzt Bernhard Küster von der TUM. Über die Hälfte der 10.000 identifizierten Proteine in den NCI-60 Zelllinien kamen in unterschiedlicher Häufigkeit in Tumor Zelllinien aller Gewebearten vor. Küster bezeichnet das entstandene Proteinportfolio als das „Kernproteom von Krebs“. Darüber hinaus beobachteten die Krebsforscher klar trennbare Proteinprofile in den verschiedeenen Tumorgeweben. Tumorzellen scheinen also die Eigenschaften gesunder Zellen des gleichen Gewebes aufzuweisen. Unter den Proteinen fanden die Forscher ganze 375 Kinasen, die für die zelluläre Kommunikation verantwortlich sind und damit beispielsweise die Zellteilung regulieren. Dieses extrem hohe Vorkommen im Vergleich zu normalen Körperzellen trägt dazu bei, dass sich Tumorzellen immer weiter vermehren, um ihr Überleben zu sichern. Darüber hinaus fanden die Forscher einen Zusammenhang zwischen Proteinmuster und der Wriksamkeit von Proteinen. „Tatsächlich gibt es Proteine, die Auskunft geben, ob eine Zelle auf bestimmte Therapeutika anspricht – oder resistent dagegen ist“, sagt Hannes Hahne von der Münchener Arbeitsgruppe. In einer Datenbank stellen die Tumorforscher ihre Proteinportfolios von NCI-60 für andere Wissenschaftler zur Verfügung. „Als nächstes werden wir das Proteom der ‚Cancer Cell Line Encyclopedia’ in Angriff nehmen, mit denen Pharmafirmen Wirkstoffe gegen Krebs testen“, so Hahne.
© biotechnologie.de/bs
MS-Therapie: Genauere Prognosen durch Biomarker
-
 <ic:message key='Bild vergrößern' />
<ic:message key='Bild vergrößern' />
- L-Selektin ist ein Zelladhäsionsmolekül, das in den Zellmembranen von Lymphozyten sitzt. Es ist an der Abwehr von Krankheitserregern beteiligt. Quelle: Pleiotrope/en.wikipedia.org
Forscher der Universität Münster haben einen Risikofaktor für eine virusvermittelte Hirnentzündung bei MS-Patienten identifiziert.
Das Medikament Natalizumab hilft besonders bei schubförmiger Multipler Sklerose, kann bei Langzeitbehandlung allerdings auch zu einer virusvermittelten Gehirnentzündung führen. Die Münsteraner Forscher haben den Grund für die Anfälligkeit der Betroffenen identifiziert und einen entsprechenden Test entwickelt. Damit ist eine individualisierte Behandlung möglich. Wie die Forscher in der Fachzeitschrift Neurology (2013, Online-Veröffentlichung) berichten, ist das Molekül L-Selektin der entscheidende Faktor. Patienten, die eine Gehirnentzündung entwickeln, fehlt das Adhäsionsmolekül offenbar.
| Mehr zum Thema auf biotechnologie.de |
News: Multiple Sklerose beginnt in der Lunge Förderbeispiel: Mehr Biomarker für die Multiple Sklerose |
L-Selektin befindet sich normalerweise auf der Oberfläche von Immunzellen und spielt eine Rolle bei Sofortreaktionen des Immunsystems auf Krankheitserreger. Um das Risiko einzuschätzen, an einer Gehirnentzündung durch die Natalizumab-Behandlung zu erkranken, wurde bisher nach Antikörpern für das Auslöservirus gesucht. Außerdem wurden Immunsuppressiva verabreicht und Wahrscheinlichkeiten berechnet. Mit dem neuen Biomarker sind wesentlich genauere Prognosen möglich. Jedem Patienten kann individuell mitgeteilt werden, ob das Risiko höher ist, eine Gehirnentzündung zu entwickeln. Der Nachweis von L-Selektin schließt die Krankheitsentstehung allerdings nicht komplett aus. „Doch der Test ist ein erster Schritt auf dem Weg, individuelle Therapie-Empfehlungen für die Natalizumab-Behandlung auszusprechen“, sagt Heinz Wiendl von der Universitätsklinik Münster. An der Publikation waren Wissenschaftler aus Osnabrück, Heidelberg und Würzburg, sowie weitere Arbeitsgruppen aus Frankreich und Italien beteiligt. In Münster erfolgte die Analyse der Blutproben von über 300 Patienten aus Europa und den USA. Die Studie entstand im Rahmen des Krankheitsbezogenen Kompetenznetzes Multiple Sklerose (KKNMS), ein Projekt das seit September 2009 vom Bundesministerium für Bildung und Forschung gefördert wird (mehr...).
© biotechnologie.de/bs