Wochenrückblick KW 32
Rückblick auf Kalenderwoche 32
Für den Zeitraum vom 4. bis 11. August 2014 hat biotechnologie.de für Sie die wichtigsten Nachrichten aus der Biotech-Branche zusammengestellt.
Forscher entschlüsseln Enzym für Safran-Aroma
-
 <ic:message key='Bild vergrößern' />
<ic:message key='Bild vergrößern' />
- Die roten Fäden, aus denen das Gewürz Safran besteht, stammen von getrockneten Blütenstempeln des Krokus Crocus sativus. Quelle: Wikipedia, CC-BY-SA-3.0
Mit der Entdeckung des Enzyms CCD2 hat ein internationales Forscherteam das Geheimnis um Aroma und Farbe des edlen Gewürzes Safran gelüftet.
Safran ist das teuerste Gewürz der Welt. Es wird aus den getrockneten Blütenstempeln der violett blühenden Krokosart Crocus sativus gewonnen. Die Pflanze wächst nur in gemäßigten Klimazonen wie Afghanistan und dem Mittelmeerraum. Ein Kilogramm der kostbaren Safranfäden kosten auf dem Markt zwischen 2.000 und 7.000 Euro. Dafür müssen 100.000 Blüten von Hand geerntet werden. Die Inhaltsstoffe dieses edlen Gewürzes chemisch zu synthetisieren, ist bisher nicht gelungen. Nun haben Forscher der Universität Freiburg gemeinsam mit Kollegen des Casaccia Research Centre in Rom sowie Wissenschaftlern aus Saudi Arabien und Spanien ein Enzym entdeckt, dass für die biotechnologische Herstellung von Safraninhaltsstoffen bedeutsam sein könnte. Die Ergebnisse der Studie stellen die Pflanzenforscher im Fachjournal PNAS (2014, Online-Vorabveröffentlichung) vor.
| Mehr auf biotechnologie.de |
News: Geheimnis um Schoko-Aroma gelüftet |
Wie das Team um Peter Beyer vom Institut für Biologie II der Universität Freiburg berichtet, ist das Enzym namens Carotenoid Cleavage Dioxygenase (kurz CCD2) sowohl für die Enstehung des bitter-herben Aroma als auch für den intensiv gelb-orangenen Farbton von Safran verantwortlich. Das Team hatte dafür die Stempel des Krokus in einer Entwicklungsphase untersucht, in der die Stoffe Crocetin/Crocins entstehen. „Wir fanden heraus, dass das Gen CCD2 in diesem Stadium besonders aktiv ist“, sagt Beyer. Danach schleusten sie das Gen in Bakterien und Maispflanzen hinein und konnten so dessen Wirkung nachweisen. Das Ergebnis: Auch in den Modellorganismen bewirkte das Enzym CCD2 eine Spaltung eines Carotinoid-Vorläufermoleküls und bewirkte damit die Synthese des Geschmacks. Safran wird aber auch als Farbstoff oder in der Medizin eingesetzt. Das Wissen um die Wirkung des Schlüsselenzymns sorgt nach Überzeugung der Forscher für ein besseres Verständnis der Biosynthese und ist damit ein erster Schritt hin zu einer biotechnologischen Herstellung von Safraninhaltsstoffen. Denn der Vorteil liegt auf der Hand. „Mit Biotechnologie ließen sich große Mengen von Crocetin/Crocins, Picrocrocin, and Safranal kostengünstig herstellen“, erklärt Beyer. Die Europäische Kommission hat die Forschung als Teil der Projekte „From DISCOvery to product“ (DISCO) und „METAPRO“ gefördert.
© biotechnologie.de/bb
Roche übernimmt dänischen RNA-Spezialisten Santaris
-
 <ic:message key='Bild vergrößern' />
<ic:message key='Bild vergrößern' />
- Roche baut seine RNAi-Techologie mit der Übernahme des dänischen Pharmaunternehmens Santaris Pharma A/S weiter aus. Quelle: Roche
Mit der Übernahme des dänischen Pharmaunternehmens Santaris Pharma für 335 Millionen Euro demonstriert Roche sein wiedererwachtes Interesse an dem Gebiet der RNAi-Technologien.
Santaris mit Sitz in Hørsholm entwickelt auf Basis der firmeneigenen Locked-Nucleic-Acid-(LNA)-Plattform neuartige RNA-basierte Medikamente. Seit Anfang des Jahres kooperieren die Dänen bereits mit Roche. Der Schweizer Pharmakonzern zahlt nun für die vollständige Übernahme des von Finanzinvestoren um Sunstone und Forbion gehaltenen dänischen Unternehmens zunächst rund 186 Millionen Euro. Beim Erreichen bestimmter erfolgsabhängiger Meilensteine könnten weitere 149 Millionen Euro hinzukommen.
„Es gibt bei vielen Krankheiten Angriffspunkte, die mit kleinen Molekülen oder Antikörpern nur sehr schwer oder überhaupt nicht erreichbar sind“, so John Reed, Leiter Forschung und frühe Entwicklung bei Roche Pharma. „Wir glauben, dass die LNA-Technologie eine Möglichkeit bietet, eine bedeutende neue Klasse von Medikamenten zu entdecken und zu entwickeln.“ Die Übernahme von Santaris ist nur die jüngste einer Reihe von kleineren Übernahmen. Seit der gescheiterten Übernahme von Illumina 2012 ist Roche auf Shoppingtour. Im Juli erst hatten die Schweizer für 540 Millionen Euro Seragon Pharmaceuticals aus San Diego erworben, einen Entwickler von Krebsarzneien. Einen Monat zuvor wurde die bis zu 257 Millionen Euro teure Übernahme des Next-Generation-Sequencing-Spezialisten Genia Technologies aus Mountain View in die Wege geleitet. Für das US-Molekulardiagnostikunternehmen Iquum Inc. kam Roche im April mit einem Angebot von bis zu 335 Millionen Euro zum Zuge.
| Mehr auf biotechnologie.de |
News: Roche investiert 90 Millionen Euro in Mannheim |
Der Zukauf des 2003 gegründeten Unternehmens Santaris markiert für Roche zudem den endgültigen Wiedereinstieg in die RNA-Interferenz (RNAi)-Forschung. 2011 hatte sich der Konzern zunächst aus dem Feld zurückgezogen, das eigene Forschungszentrum in Kulmbach im Rahmen eines Management-Buyouts abgestoßen. Anfang 2013 schließlich die Wende: Mit dem US-amerikanischen Antisense-Spezialisten Isis Pharmaceuticals wurde eine Forschungsallianz für Chorea Huntington geschmiedet. Im Januar 2014 folgte dann ein erster Lizenzvertrag zwischen Roche und Santaris. Für die Entwicklung von Antisense-Arzneien auf Basis der LNA-Plattform stellte der Pharmakonzern dem dänischen Biotech-Unternehmen eine Sofortzahlung von rund 7,5 Millionen Euro sowie Meilensteine im Wert von rund 102 Millionen Euro in Aussicht. Dies Geld dürfte nun in jedem Fall im Konzern bleiben – als Roche Innovation Center Kopenhagen gehört Santaris künftig direkt zum Roche-Firmenverbund.
© biotechnologie.de/bk
Winzige Mikroben-WGs im Erdöl
-
 <ic:message key='Bild vergrößern' />
<ic:message key='Bild vergrößern' />
- Der Pitch Lake in Trinidad ist ein Asphaltsee: Hier steigt flüssiges Öl an die Oberfläche auf. Trotzdem bieten sich hier Lebensräume. Quelle: Rainer Meckenstock
Winzige Wassertropfen in Erdöl beherbergen eine verblüffende Vielfalt an Mikroben. In den Tröpfchen mit einem Volumen von einem Mikroliter eingeschlossen helfen sie beim Abbau des Öls.
Das haben Wissenschaftler des Helmholtz Zentrums München bei Studien am Pitch Lake, einem Asphaltsee, herausgefunden. Sie berichten im Fachjournal Science (2014, Bd.345, Nr.6197, S.673). Erdöl klingt zunächst eher nach lebensfeindlichem Terrain. Innerhalb des Öls vom Pitch Lake jedoch befinden sich winzige, suspendierte Wassertröpfchen. „Darin haben wir komplexe mikrobielle Lebensgemeinschaften gefunden, die dort aktiv das Öl abbauen“, sagt Rainer Meckenstock vom Helmholtz Zentrum München. Bisher ging man davon aus, dass der mikrobielle Ölabbau nur an der Übergangszone zwischen reinen Öl- und Wasserphasen stattfindet. Der Asphaltsee Pitch Lake auf der Karibikinsel Trinidad liegt über einer geologischen Besonderheit: Durch einen Riss in der Erdkruste strömt Öl, das in etwa 1000 Metern Tiefe unter großem Druck lagert, nach oben. Der See bietet eine der seltenen Möglichkeiten, natürlich aufgestiegenes Öl zu analysieren, etwa auf die Existenz von Mikrobenpopulationen. An klassischen Förderanlagen können Wissenschaftler nur Öl untersuchen, das hohem Druck ausgesetzt und mit Wasser und anderen Stoffen verunreinigt ist.
| Mehr auf biotechnologie.de |
Am Pitch Lake wird das Öl bei seinem Aufstieg zu natürlichem Asphalt. Darin eingeschlossen fanden die Forscher winzige Wassertröpfchen, mit komplexen Lebensgemeinschaften aus mehr als 20 verschiedeneren Mikroben, darunter Archeen und Bakterien. Das Team um Meckenstock hat mit Kollegen der Technischen Universität Berlin, der Washington State University und University of West Indies konnte damit nachweisen, dass auch innerhalb der Ölphase Abbauprozesse erfolgen. „Der Abbau verändert die chemische Zusammensetzung des Erdöls, schlussendlich kann zähflüssiger Bitumen entstehen, wie in Ölsanden“, erklärt Meckenstock. „Unsere Daten liefern also wichtige Informationen für die Qualität von Erdöl und sind daher essenziell für die Industrie rund um die weltweit noch immer wichtigste Energiequelle.“ Was dem Öl schadet, nämlich der Abbau seiner chemischen Verbindungen (Kohlenwasserstoffe), kann in kontaminiertem Grundwasser durchaus erwünscht sein. Die Mikroorganismen, die an einen extrem toxischen Lebensraum angepasst sind, könnten neue Konzepte für den Schadstoffabbau im Grundwasser eröffnen.
© biotechnologie.de/pg
Protein-Parcours durch die Zelle simuliert
-
 <ic:message key='Bild vergrößern' />
<ic:message key='Bild vergrößern' />
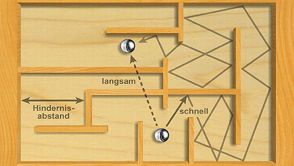
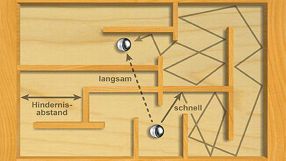
- Ein Protein tastet sich im Stop-and-go-Modus durch das innere Labyrinth der Zelle, so haben es die Heidelberger Bioinformatiker modelliert. Quelle: Karsten Rippe
Membranen, das Zytoskelett und das Erbgut im Zellkern bilden ein dynamisches dreidimensionales Labyrinth innerhalb einer Zelle. Diese Hindernisse bremsen die Bewegungen von Proteinen. Wissenschaftler vom BioQuant-Zentrum der Universität Heidelberg und vom Deutschen Krebsforschungszentrum (DKFZ) ist es gelungen, die Zelltopologie zu „kartieren“, indem sie Proteine in der lebenden Zelle über verschiedene Zeitspannen und Distanzen beobachtet haben. Zum Einsatz kam dabei ein neues Verfahren, das auf Fluoreszenzmikroskopie basiert. Damit konnten die Wissenschaftler messen, wie lange Proteine brauchen, um im Zellinneren Strecken in einem Bereich von 0,2 bis 3 Mikrometern zurückzulegen. Aus der Analyse der Messdaten entwickelte das Team unter der Leitung von Karsten Rippe ein mathematisches Modell, mit dem sich intrazelluläre Strukturen rekonstruieren lassen. Die Forschungsergebnisse wurden im Fachjournal Nature Communications (2014, Online-Veröffentlichung) publiziert. „Bisher war unklar, wie ein in der Zelle diffundierendes Protein das innere Netzwerk von Hindernissen erspürt“, erläutert Rippe. Sein Team hat eine Methode entwickelt, mit der die räumliche Struktur in einer Zelle aus zufälligen Proteinbewegungen abgeleitet werden kann.
| Mehr auf biotechnologie.de |
„Die Bewegungen eines Proteins in der Zelle ähneln denen einer Kugel in dem Geschicklichkeitsspiel, bei dem eine Murmel durch ein Labyrinth ,bugsiert‘ werden muss“, sagt der Co-Autor der Studie, Michael Baum. Die Murmeln können leicht über kurze Strecken bewegt werden, dann kollidieren sie jedoch mit einem Hindernis und werden auf ihrem weiteren Weg gebremst. Das führt dazu, dass sie sich über längere Strecken mit einer verlangsamten Geschwindigkeit per „stop and go“ fortbewegen. Die Forscher schufen ein mathematisches Modell, mit dem sich die gemessene Bewegung der Proteine in der Zelle beschreiben und die Zelltopologie rekonstruieren lässt – und zwar mit einer deutlich besseren Auflösung als es aktuell mit einer lichtmikroskopischen Abbildung möglich ist. „Die Struktur der Hindernisse, auf die ein Protein bei seiner Bewegung durch die Zelle trifft, ähnelt dem Modell eines löchrigen Mediums, wie es zum Beispiel auch in einem Schwamm zu finden ist“, sagt der Heidelberger Wissenschaftler. In dieser dynamischen Struktur waren größere Proteine zuweilen mehrere Minuten lang gefangen. Die Forschungsarbeiten wurden vom Bundesministerium für Bildung und Forschung im Rahmen der Initiativen e:Bio und SysTec unterstützt.
© biotechnologie.de/pg